Visualisering av samband mellan variabler
Spridningsdiagram
När ett datamaterial innehåller flera variabler kan det vara intressant att undersöka vilka (om några) variabler har ett samband med varandra. Detta kan göras på olika sätt, men visualisering i ett spridningsdiagram är ett sätt som möjliggör att se många olika typer av samband mellan två variabler.
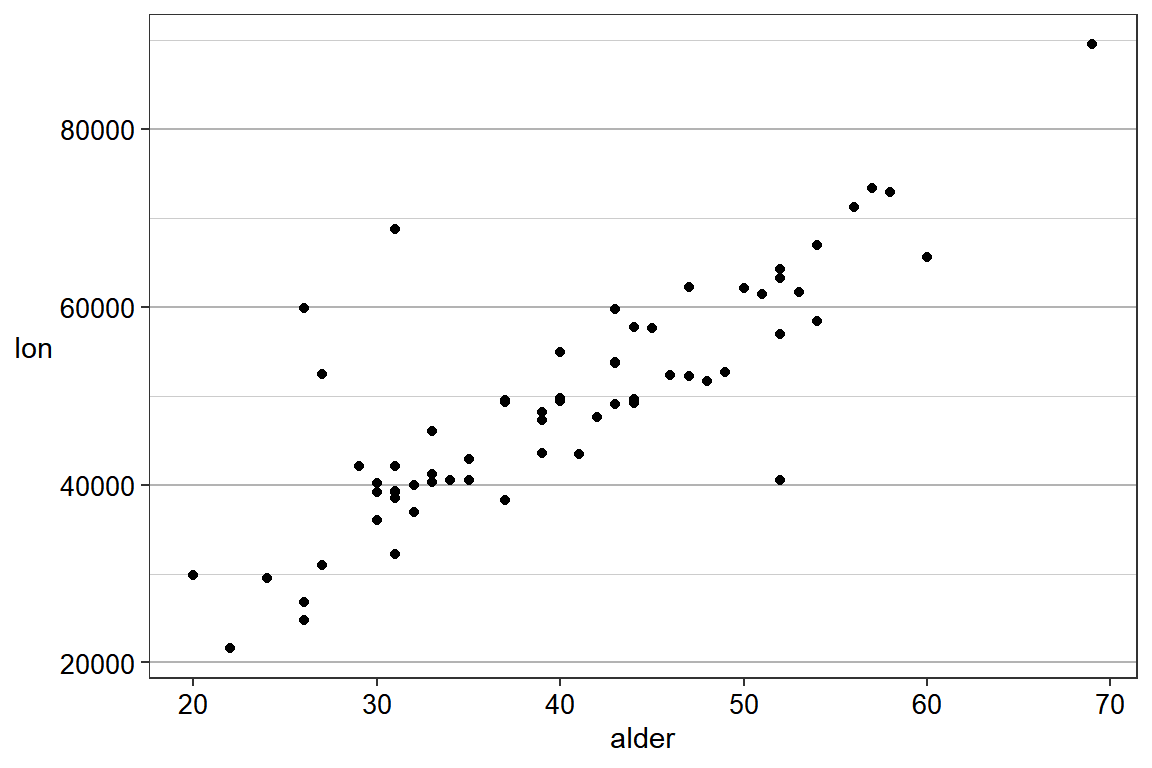
Vi utgår ifrån tidigare datamaterial och fokuserar på de två kvantitativa variablerna alder och lon i aes(). Här bör vi välja variabler till de olika axlarna som medför en logisk tolkning av vilken variabel som förklarar den andra. Den förklarande variabeln, x, anser vi förklara responsvariabeln, y, och med dessa variabler är det mer logiskt att alder förklarar lon för en individ.
För att skapa ett spridningsdiagram används geom_point(). Argument som kan vara av intresse i denna funktion är color, shape eller size.
p <- ggplot(exempeldata) + aes(x = alder, y = lon) + geom_point()
p
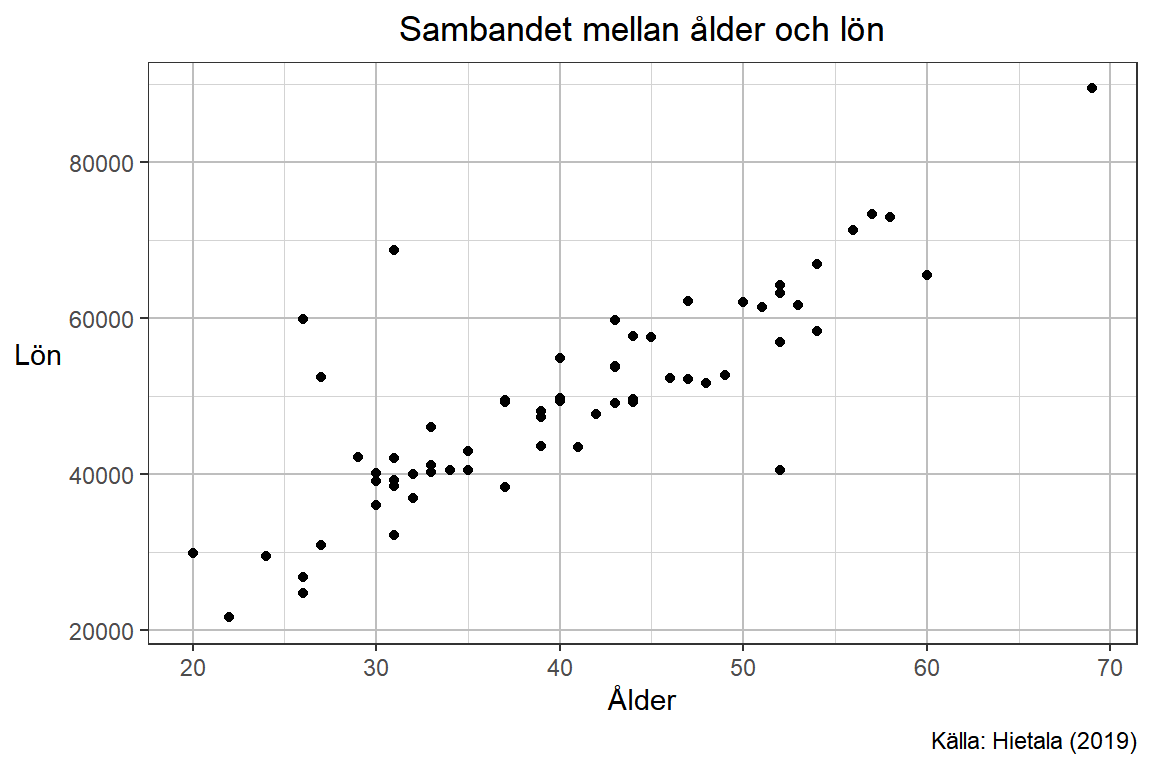
Som vanligt lägger vi till lite extra kod för att ändra utseendet av diagrammet. När det kommer till stödlinjer och spridningsdiagram är syftet med diagrammet att se helheten, det generella sambandet mellan variablerna, mer än specifika värden för enstaka observationer. Detta betyder att stödlinjerna riskerar att ta för mycket fokus från diagrammet istället för att tillföra tydlighet. En lösning på detta är att försvaga styrkan på linjerna eller helt ta bort dem.
p <-
p + theme_bw() +
theme(axis.title.y =
element_text(angle = 0,
hjust = 1,
vjust = 0.5),
plot.title =
element_text(hjust = 0.5),
plot.subtitle =
element_text(hjust = 0.5),
panel.grid.major.x =
element_line(color = "gray"),
panel.grid.minor.x =
element_line(color = "light gray"),
panel.grid.major.y =
element_line(color = "gray"),
panel.grid.minor.y =
element_line(color = "light gray")) +
labs(title = "Sambandet mellan ålder och lön",
x = "Ålder",
y = "Lön",
caption = "Källa: Hietala (2019)")
p
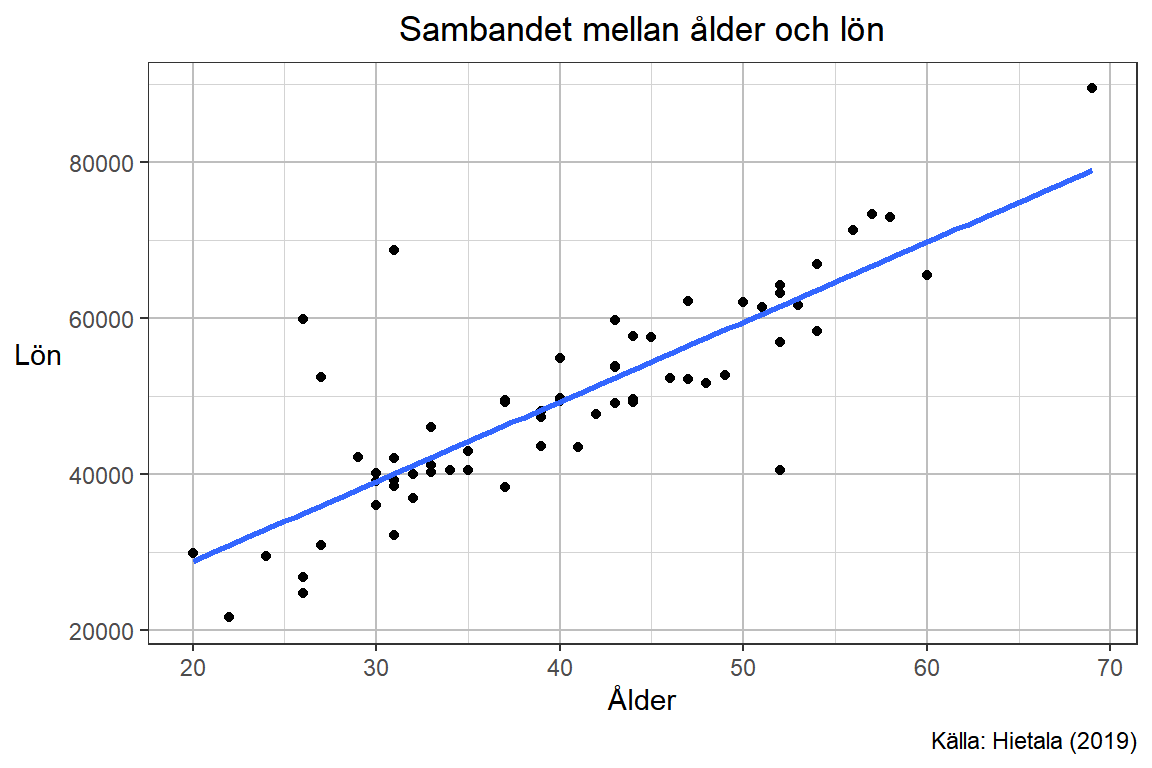
Då vi pratar om samband är det naturligt att gå vidare till en statistisk metod som kallas för regression som mer i detalj beskriver sambandet mellan variabler. Vi kommer inte gå igenom det så mycket i denna kurs men vi kan fortfarande visualisera den skattade regressionslinje som fås med hjälp av en metod som kallas minsta kvadrat skattning. I R lägger vi till ett till geom-objekt till samma diagram. method = lm anger att vi vill skatta en linjär modell (linear model) och se = FALSE anger att vi inte vill visa s.k. konfidensband.
p + geom_smooth(method = lm, se = FALSE)
Visualisering av olika datamaterial i samma diagram
Om extremvärden skulle finnas i datamaterialet kommer sambandet (och en inritad regressionslinje) riskera att förskjuta det generella samband som finns. Observera att det i praktiken inte är så lätt att bara plocka bort extremvärden från datamaterialet utan en djupare dykning i orsakerna till detta värde. Är extremvärden en felinmatning eller är det ett riktigt värde som är en del av målpopulationen? För att visa hur vi kan använda olika datamaterial i samma diagram kommer vi göra det naïva och plocka bort dessa.
I detta skede av er utbildning rekommenderas att ni identifierar dessa punkter i diagrammet och manuellt tar bort dessa ur data i Excel, sparar en ny fil med nytt namn, och importerar detta nya material till R och sparar det som ett nytt objekt.
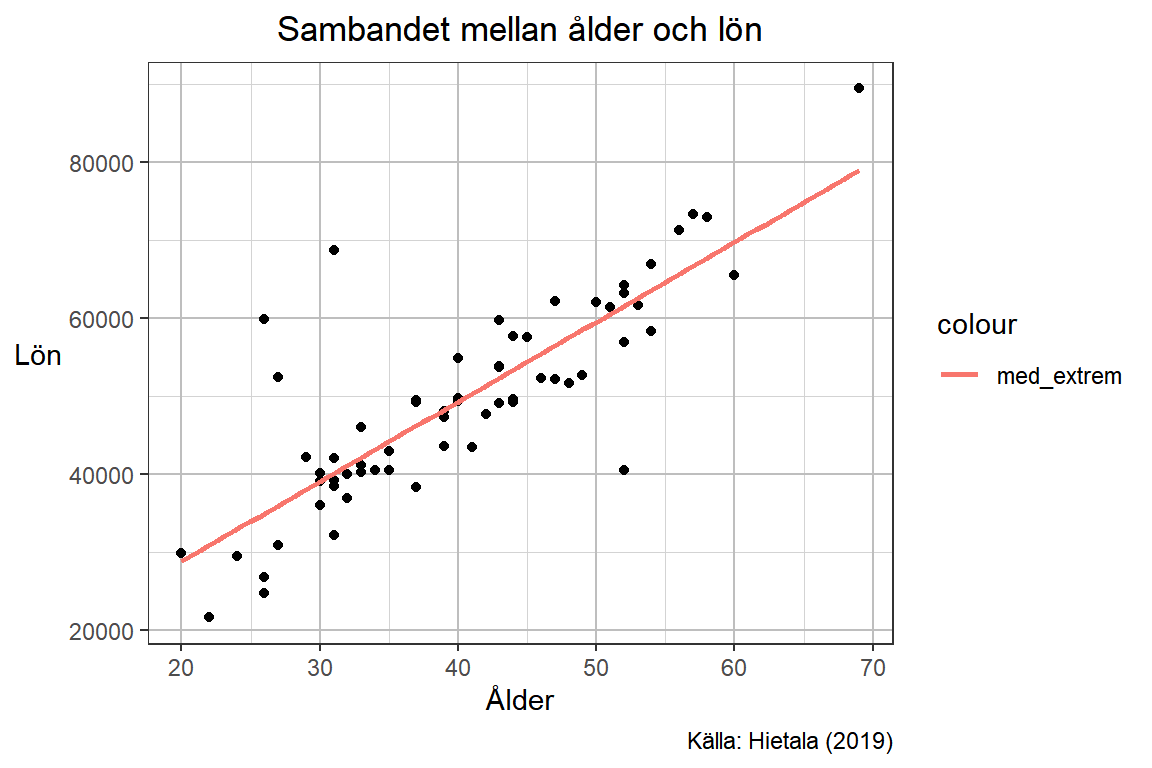
Det första vi måste göra för att diagrammet ska bli tydligt är att skapa en grupp (och tillhörande legendrad) för vardera regressionslinje. Detta görs genom att använda aes(color = "gruppnamn") inuti geom_smooth(). Vi ser nu att en legend lagts till i diagrammet med det angivna namnet på linjen som vi skrivit.
p <-
p + geom_smooth(aes(color = "med_extrem"),
method = lm,
se = FALSE)
p
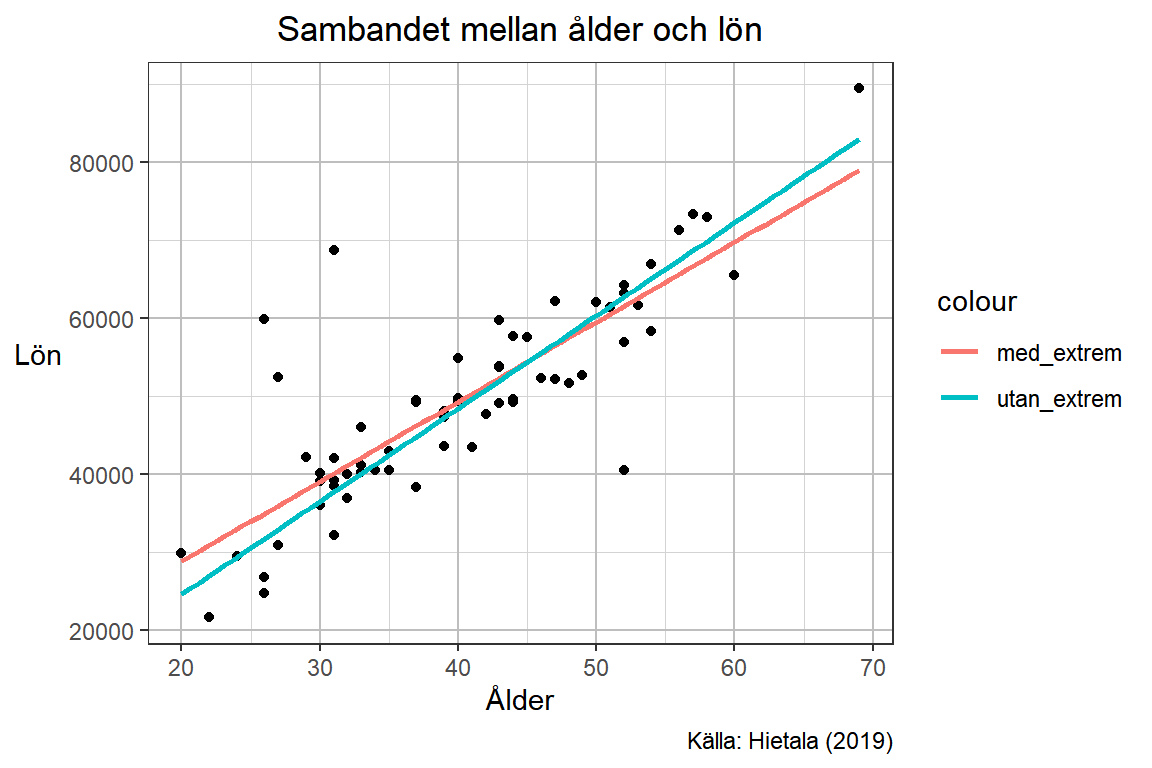
För att lägga till den nya regressionslinjen utan extremvärden anges i geom_smooth() ett nytt data-objekt. Linjen kommer då ritas med samma variabler som angivits innan men utgå från detta materials värden, som saknar de extremvärden som man kan urskilja i materialet.
p <-
p + geom_smooth(data = exempeldata_extrem,
aes(color = "utan_extrem"),
method = lm,
se = FALSE)
p
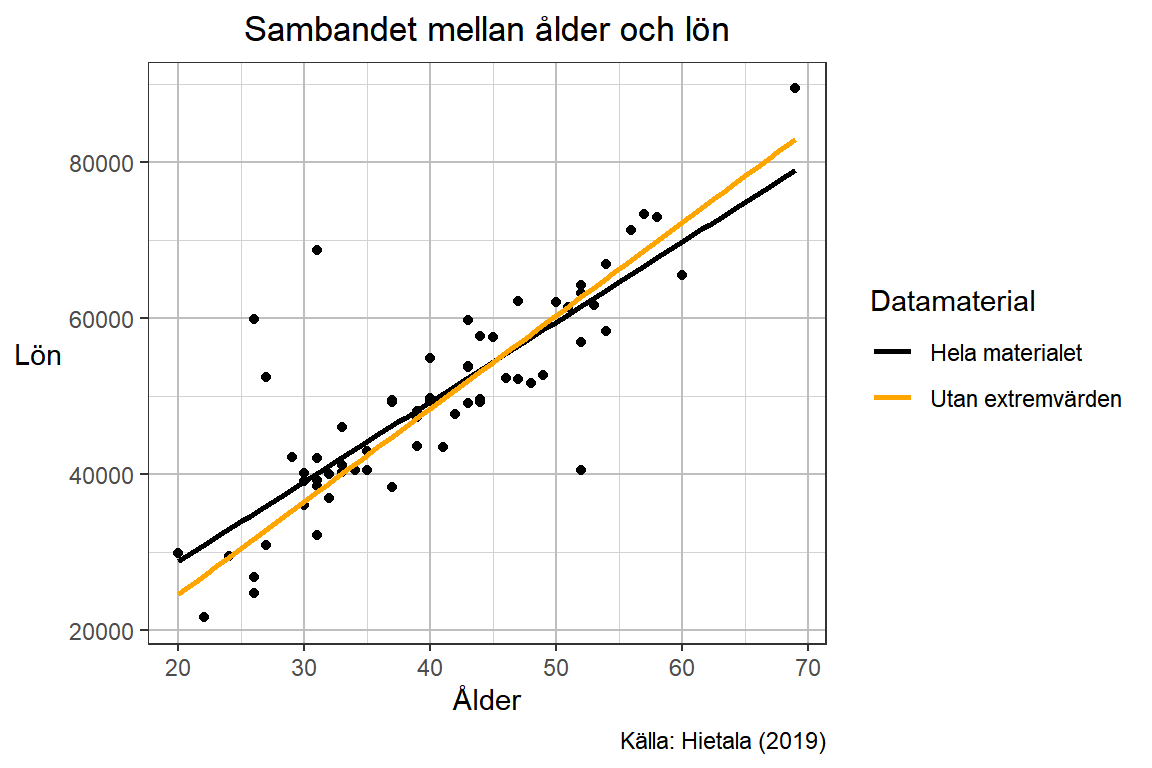
Legenden kan behöva förtydligas lite och här får vi återigen användning av de gruppnamn som angetts tidigare i koden. Argumentet name ger en bättre titel på legenden och i values kan vi i vektorn säga specifikt vilka grupper som vi vill ha vissa färger likt "gruppnamn" = "färg". Vi vill också ange bättre etiketter på dessa grupper och detta görs med labels-argumentet.
p <-
p + scale_color_manual(name = "Datamaterial",
values = c("med_extrem" = "black",
"utan_extrem" = "orange"),
labels = c("med_extrem" = "Hela materialet",
"utan_extrem" = "Utan extremvärden"))
p
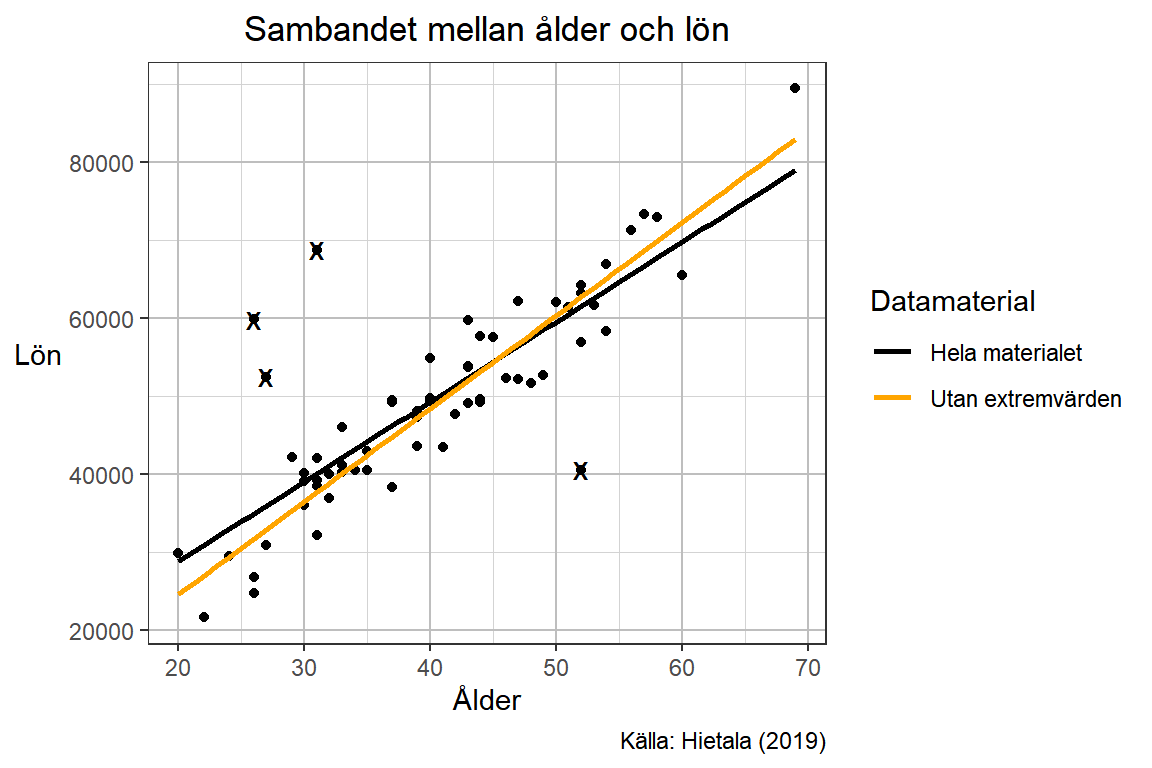
Extra visualisering
Det kanske också kan vara intressant att peka ut vilka observationer som plockas bort vilket kan göras genom att skapa ytterligare ett datamaterial med enbart de valda extremvärdena. Vi kan då lägga till ett till geom_point() till diagrammet där vi ändrar shape till någon annan symbol som tydliggör att dessa har plockats bort.
p + geom_point(data = exempeldata_extrem_points,
shape = "x",
size = 4)
Linjediagram
För att vi ska kunna skapa ett linjediagram över flera serier behöver vi importera data med grupperingskolumner. Även om vi enbart ska visualisera en serie är det bra att få in rutinen att representera data i R på detta sätt.
Datamaterialet bör alltså se ut som följer:
| År | Region | Antal |
|---|---|---|
| 1975 | Hela landet | 331 |
| 1976 | Hela landet | 332 |
| 1977 | Hela landet | 371 |
| 1978 | Hela landet | 364 |
| 1979 | Hela landet | 364 |
När materialet nu innehåller tre variabler (en som visar tid, en som visar vilken grupp värden och år tillhör, samt mätvärdet för den angivna gruppen och året) kan vi påbörja visualiseringen.
Exempeldata innehåller information om antalet anmälda våldsbrott per 100 000 invånare i hela landet, Västernorrland och Östergötland och är hämtat från SCB.
En tidsserie
Om endast en serie ska visualiseras kan vi plocka ut en grupp från det tidigare materialet och visualisera endast det. Vi gör detta med filter() från dplyr-paketet. Kodexemplet gör denna filtrering inuti ggplot() men vi skulle lika gärna skapa ett filtrerad data som sparas som ett nytt objekt och använda det senare i ggplot().
## Anger att vi endast vill ha en del av data i diagrammet
p <-
ggplot(
filter(.data = tidsserie_exempel, Region == "Hela landet")
)
När vi nu sagt att endast en del av materialet ska visualiseras kan vi som tidigare ange aes() där vi nu måste ange både x och y. På x-axeln vill vi ha tiden, alltså År i vårt exempelmaterial, och y-axeln ska innehålla mätvärden, i detta fall variabeln Antal.
För att skapa ett linjediagram används geom_line(). I den funktionen finns argument såsom color, linetype, size och liknande som kan användas för att ändra utseendet av diagrammet. Ibland kan linjen bli väldigt smal och svår att se men då kommer argumentet size väl till pass.
p <-
p + aes(x = År, y = Antal) +
geom_line(color = "dark orange", size = 1)
p
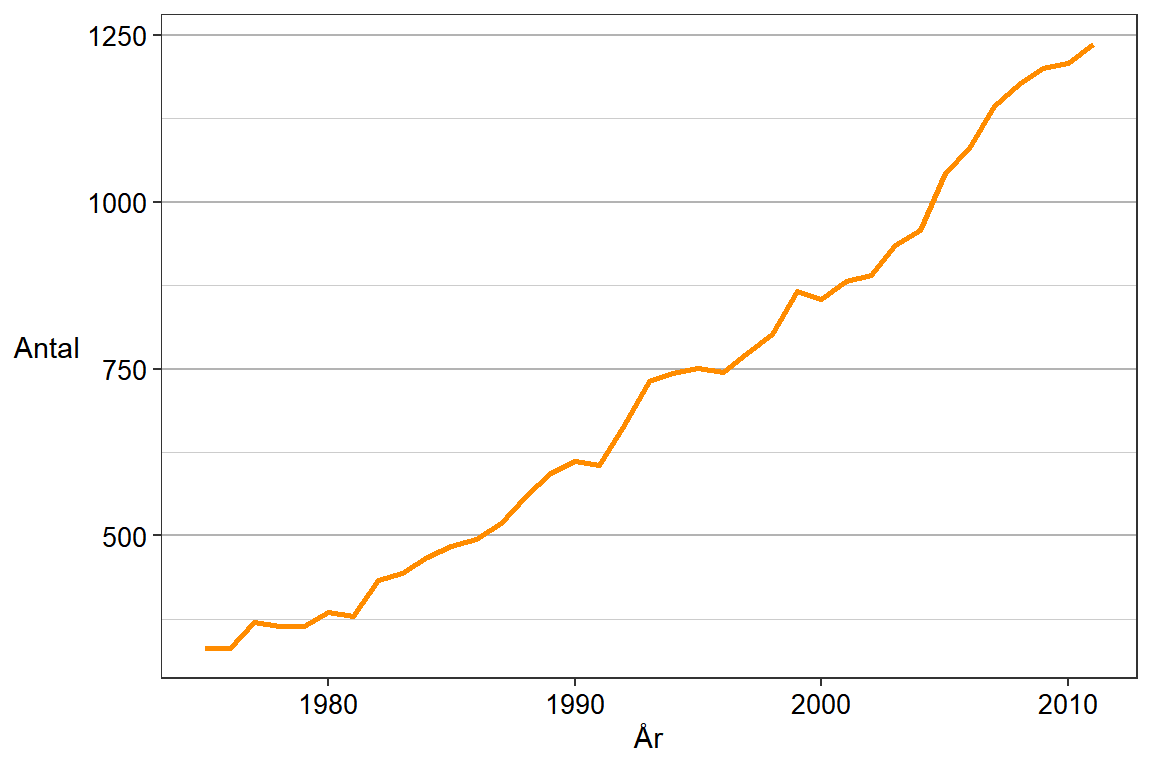
Som tidigare ändrar vi det visuella och lägger till ytterligare förtydligande texter med liknande koder som innan. Till linjediagram vill vi ofta kunna se både lod- och vågräta avstånd vilket innebär att stödlinjer bör finnas åt båda hållen. Det kan dock vara av intresse att använda och ändra på panel.grid.minor-linjerna till en svagare färg för att göra skillnad på de olika stödlinjerna. I exemplet nedan används en mörkare grå färg för panel.grid.major.
Något som också introducerats i nedanstående kod är plot.subtitle i theme() som styr egenskaper av undertiteln som i detta diagram kan vara bra för att förtydliga vilken region som materialet visar. Undertitelns text läggs till i labs() och subtitle-argumentet.
p <-
p + theme_bw() + theme(axis.title.y =
element_text(angle = 0,
hjust = 1,
vjust = 0.5),
plot.title =
element_text(hjust = 0.5),
plot.subtitle =
element_text(hjust = 0.5),
panel.grid.major.x =
element_line(color = "dark gray"),
panel.grid.minor.x =
element_line(color = "gray"),
panel.grid.major.y =
element_line(color = "dark gray"),
panel.grid.minor.y =
element_line(color = "gray")) +
labs(y = "Antal",
x = "År",
title = "Antal anmälda våldsbrott per 100 000 invånare",
subtitle = "Hela landet",
caption = "Källa: SCB (2012)")
p
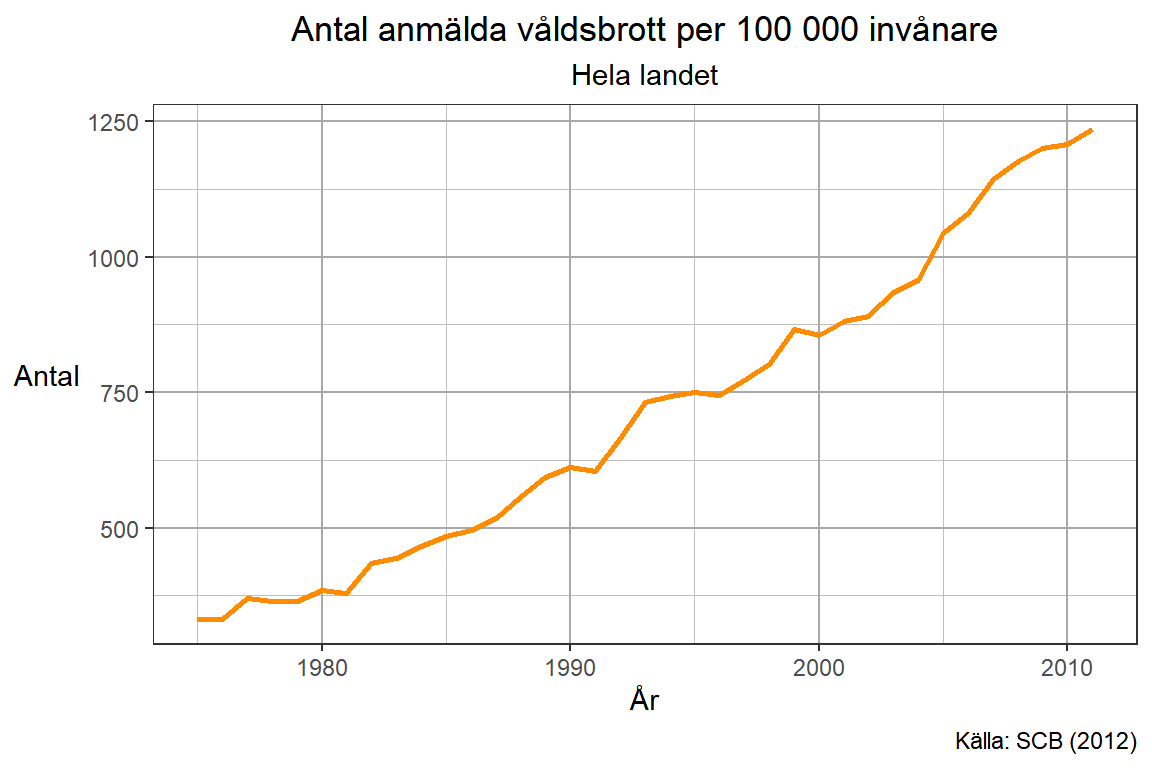
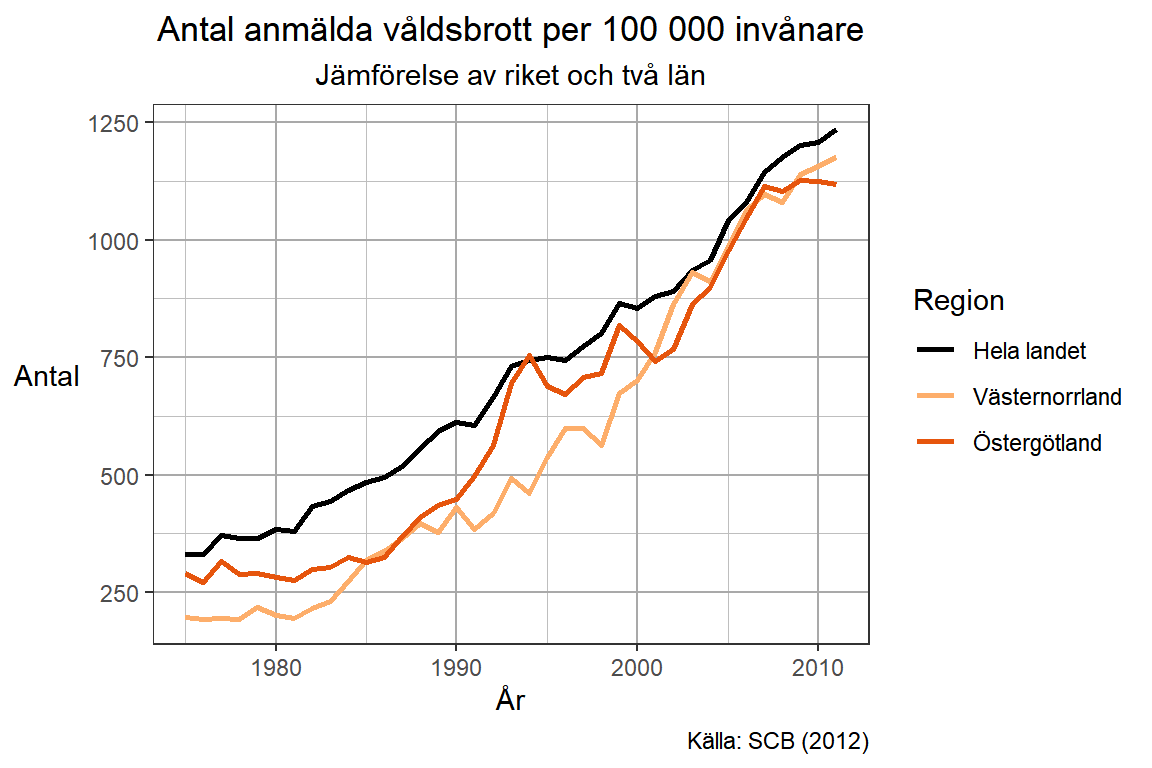
Flera tidsserier
För att visualisera flera tidsserier i ett diagram kräver ggplot2 att datamaterialet ska vara formaterad med en grupperingsvariabel. För att R ska göra skillnad på dessa olika grupper måste group och/eller color argumentet i aes innehålla den grupperingsvariabel som finns i data.
p <- ggplot(tidsserie_exempel) +
aes(x = År,
y = Antal,
color = Region,
group = Region) +
geom_line(size = 1)
p
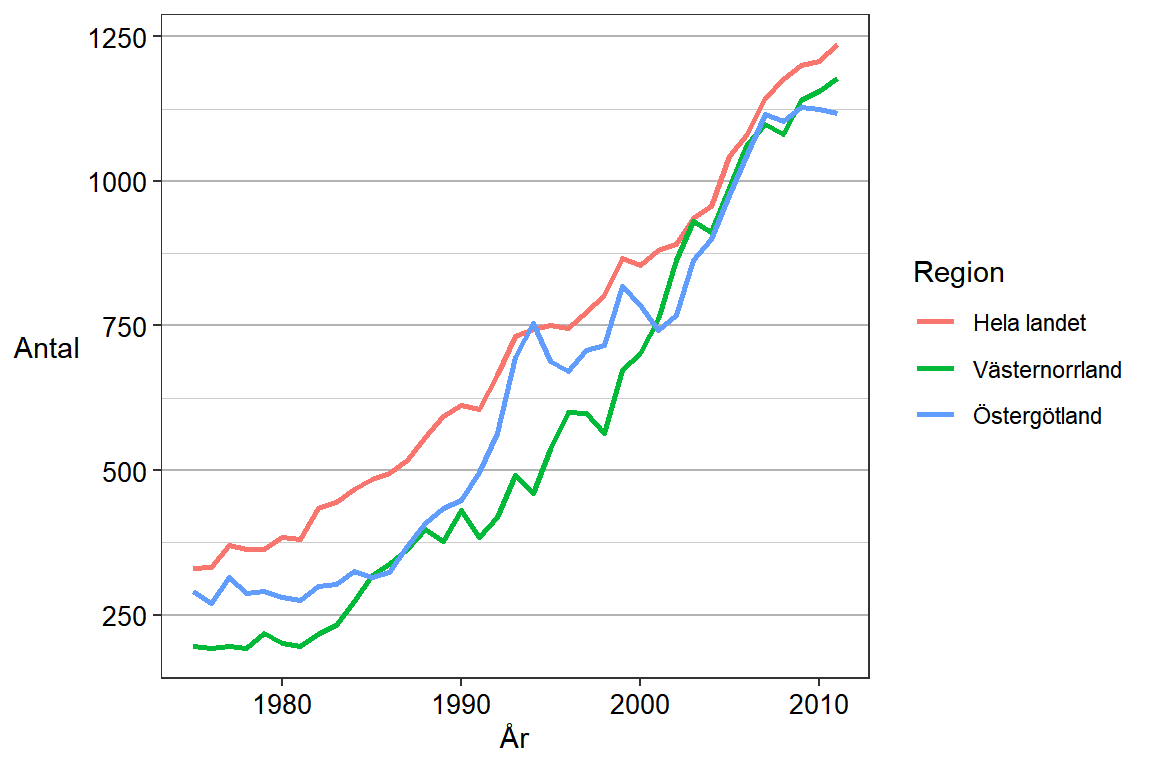
Som tidigare kan vi nu ändra om flera aspekter av diagrammet men nu måste vi också ändra den legend som skapas från grupperna i aes(). När vi nu vill ändra linjefärger är det funktionen scale_color_manual() som ska användas, specifikt argumentet values.
Problemet som vi kommer märka är att det är svårt att skapa en vektor med färger som är tydliga nog för olika situationer. Kodexemplet nedan försöker använda delar av brewer.pal(palette = "Oranges") tillsammans med en svart grundfärg för att bibehålla en färgstil.
brewer.pal()-funktionen försöker plocka färger från den angivna paletten som skiljer sig från varandra men att ange n = 3 för de tre regionerna ger en alltför ljus färg som den första. Indexeringen [-1] plockar bort den första ljusa färgen och vi lägger istället till svart först i values-vektorn.
p <-
p + scale_color_manual(values =
c("black", brewer.pal(n = 3, "Oranges")[-1])) +
theme_bw() + theme(axis.title.y =
element_text(angle = 0,
hjust = 1,
vjust = 0.5),
plot.title =
element_text(hjust = 0.5),
plot.subtitle =
element_text(hjust = 0.5),
panel.grid.major.x =
element_line(color = "dark gray"),
panel.grid.minor.x =
element_line(color = "gray"),
panel.grid.major.y =
element_line(color = "dark gray"),
panel.grid.minor.y =
element_line(color = "gray")) +
labs(y = "Antal",
x = "År",
title = "Antal anmälda våldsbrott per 100 000 invånare",
subtitle = "Jämförelse av riket och två län",
caption = "Källa: SCB (2012)")
p