Klustring
Klustringmetoder ämnar att hitta underliggande grupperingar av observationer (eller variabler) i datamaterialet som inte är angivna från början, ett exempel på oövervakad inlärning. I verkliga fall är det väldigt ovanligt att materialet kan visualiseras och man med blotta ögat kan identifiera dessa grupperingar, men i detta underlag kommer första steget av varje algoritm innehålla ett visualiseringssteg för att kunna påvisa vilka algoritmer som klarar av att hitta vilka sorters klustertyper.
Såvida inte skalan på variablerna är av specifikt intresse att ta hänsyn till vid tolkningen, ska alltid variablerna standardiseras för att låta varje variabel bidra lika mycket till avståndsberäkningar.
Vad är ett kluster?
Ett kluster definieras som en grupp av enheter vars avstånd till varandra är mindre än avståndet till en annan grupp av enheter. Alltså homogena grupper där variationen inom gruppen är liten och variationen mellan grupperna är stor.
K-means
Den enklaste och effektivaste algoritmen är K-means.
## Visualisera data
ggplot(data = data_clust_circ, aes(x = X, y = Y)) + geom_point() + theme_bw() +
theme(axis.title.y = element_text(angle = 0, vjust = 0.5))
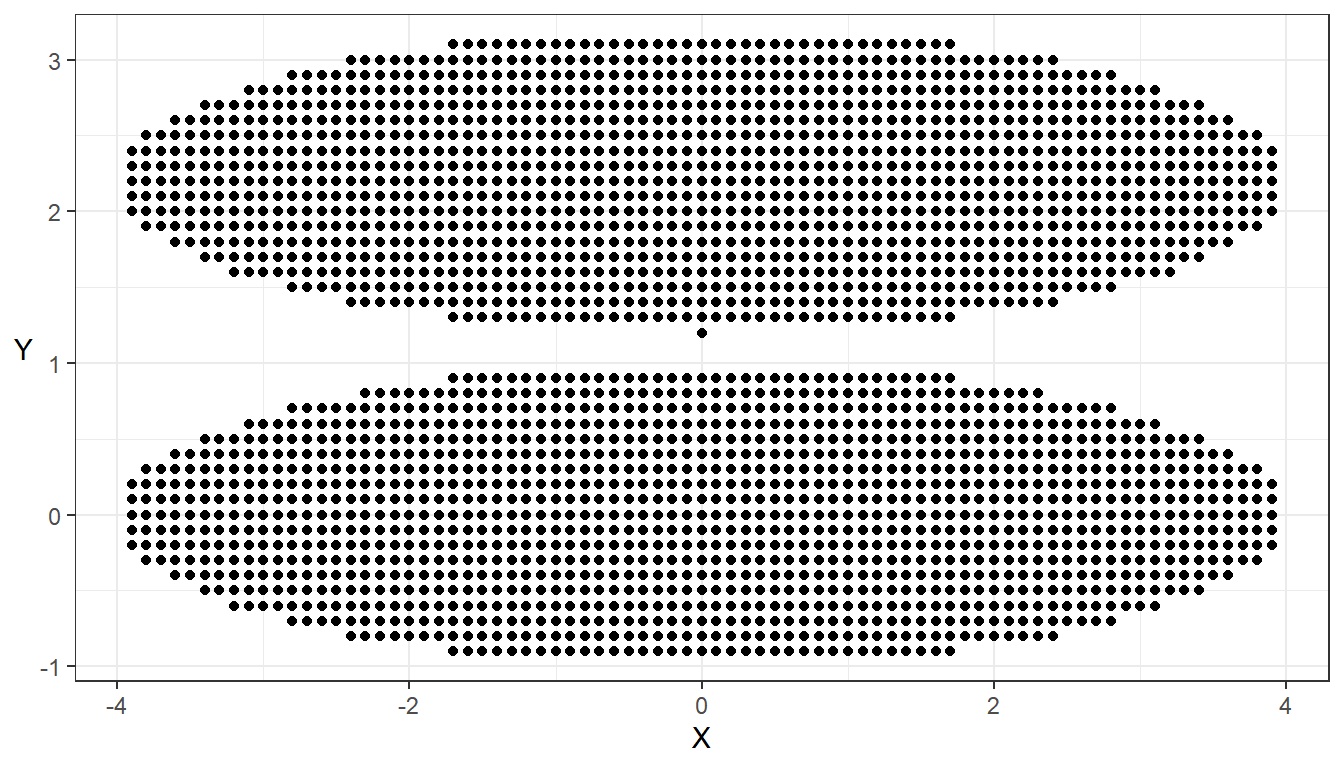
## STANDARDISERA
data_clust_circ_std <- data.frame(scale(data_clust_circ, center = TRUE, scale = TRUE))
## Visualisera
ggplot(data = data_clust_circ_std, aes(x = X, y = Y)) + geom_point() + theme_bw() +
theme(axis.title.y = element_text(angle = 0, vjust = 0.5))
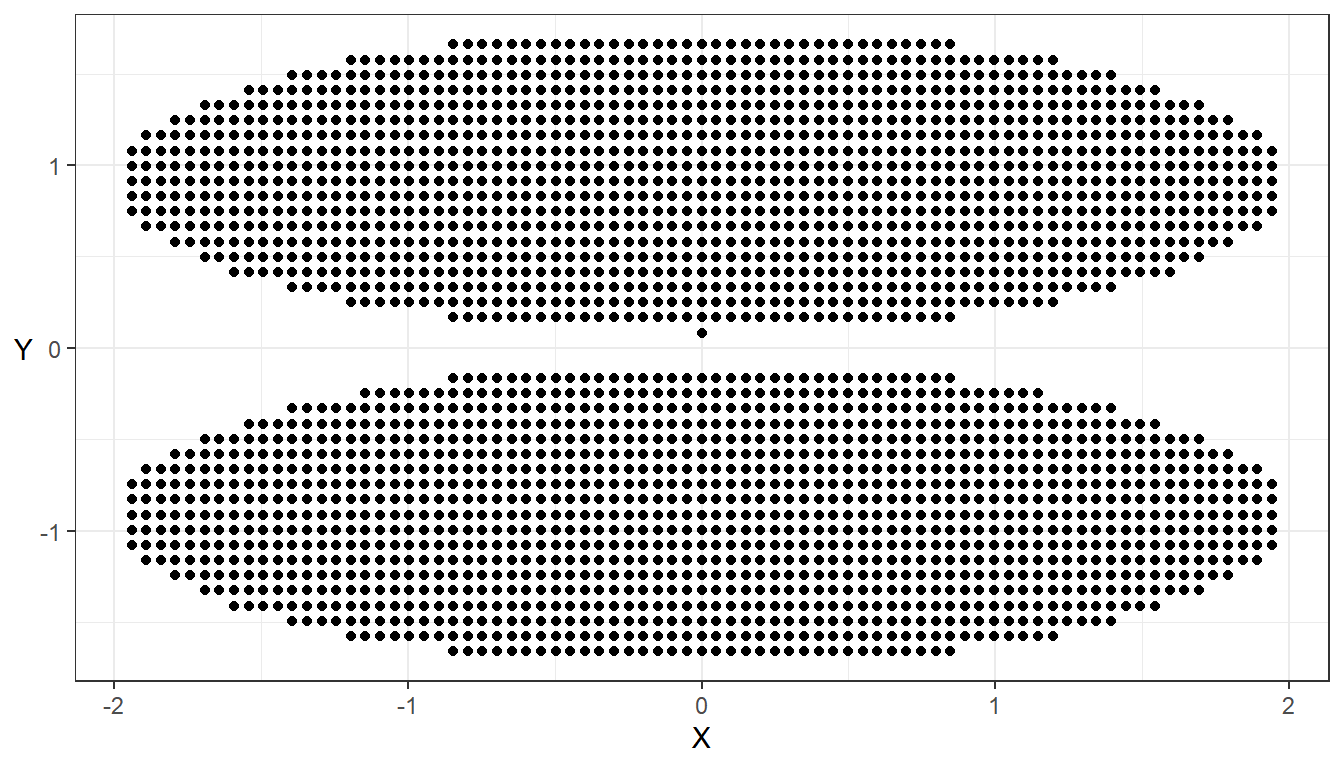
## Kör K-means algoritmen
cluster_result <-
kmeans(
x = data_clust_circ_std,
# Antalet centroider (k). Om man vill ange sina egna centroider rbind(c(x1, y1), c(x2, y2), ...)
centers = 2,
# Antalet maximala iterationer som algoritmen tillåts att köra innan den slutar
iter.max = 1,
# Antalet set av startcentroider som undersöks. Endast aktiv om centers = k
nstart = 1
)
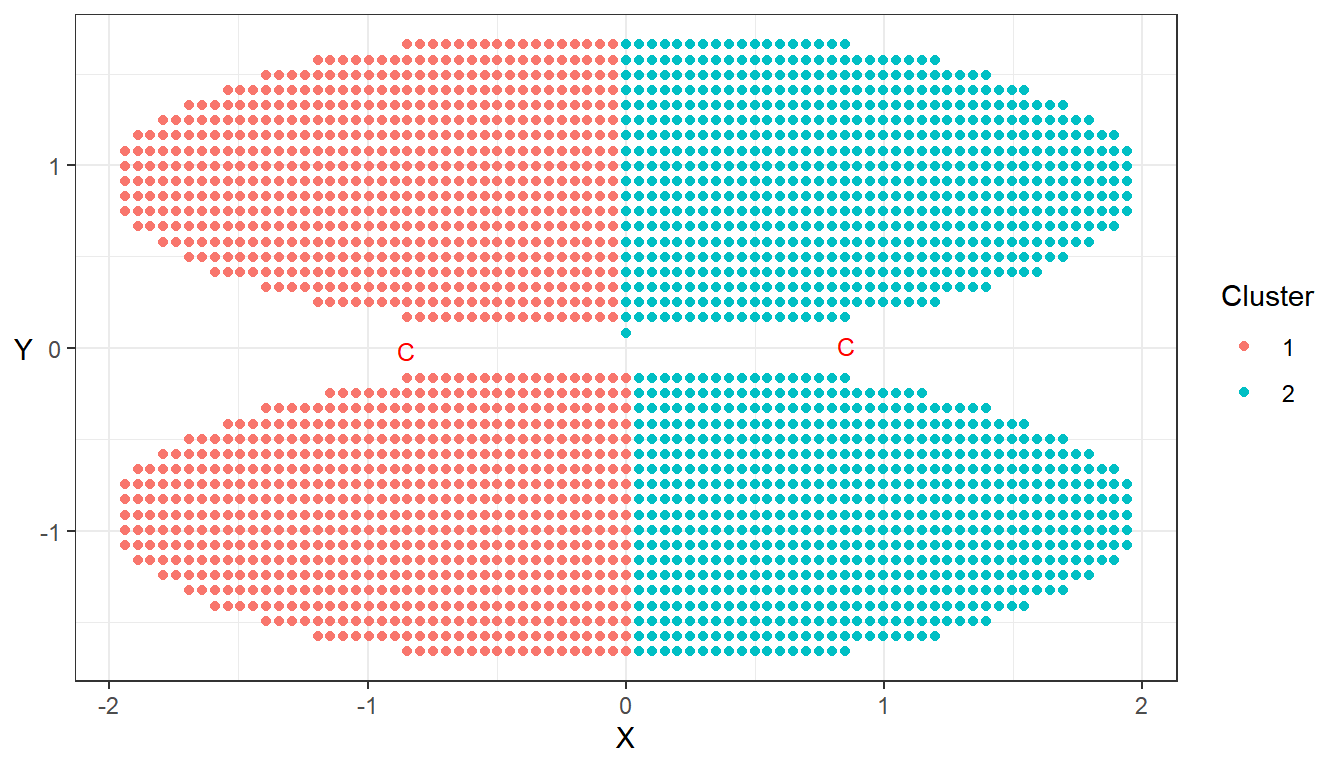
I resultatet av kmeans() sparas dels klustertillhörigheten när konvergens har uppnåtts (eller att det angivna maximala antalet iterationer har körts). Vi kan använda $ för att plocka ut enskilda delar av detta objekt, exempelvis $centers för centroidernas koordinater som kan användas för visualisering av klusterresultatet.
## Visualiserar resultatet
ggplot(data = data_clust_circ_std, aes(x = X, y = Y)) +
# Ritar punkterna enligt deras klustertillhörighet
geom_point(aes(color = factor(cluster_result$cluster))) + theme_bw() +
# Ritar ut klustercentroiderna i diagrammet
geom_point(data = data.frame(cluster_result$centers),
mapping = aes(x = X, y = Y),
shape = "C",
size = 3,
color = "red") +
theme(axis.title.y = element_text(angle = 0,
vjust = 0.5)) +
scale_color_discrete(name = "Cluster")
Hierarkisk klustring
En mer flexibel metod som kan ta hänsyn till olika distansmått och länkningsmetoder. Agglomerativ hierarkisk klustring är den metod som börjar med alla observationer i enskilda kluster och de närmaste klustren kontinuerligt slås ihop tills vi når ett kluster. I R använder vi funktionen agnes() för denna algoritm.
Ett alternativ är att använda Divisive Hierarchical Clustering som går åt andra hållet, alltså börjar med alla observationer i ett kluster och delar upp kluster tills alla observationer är i enskilda kluster.
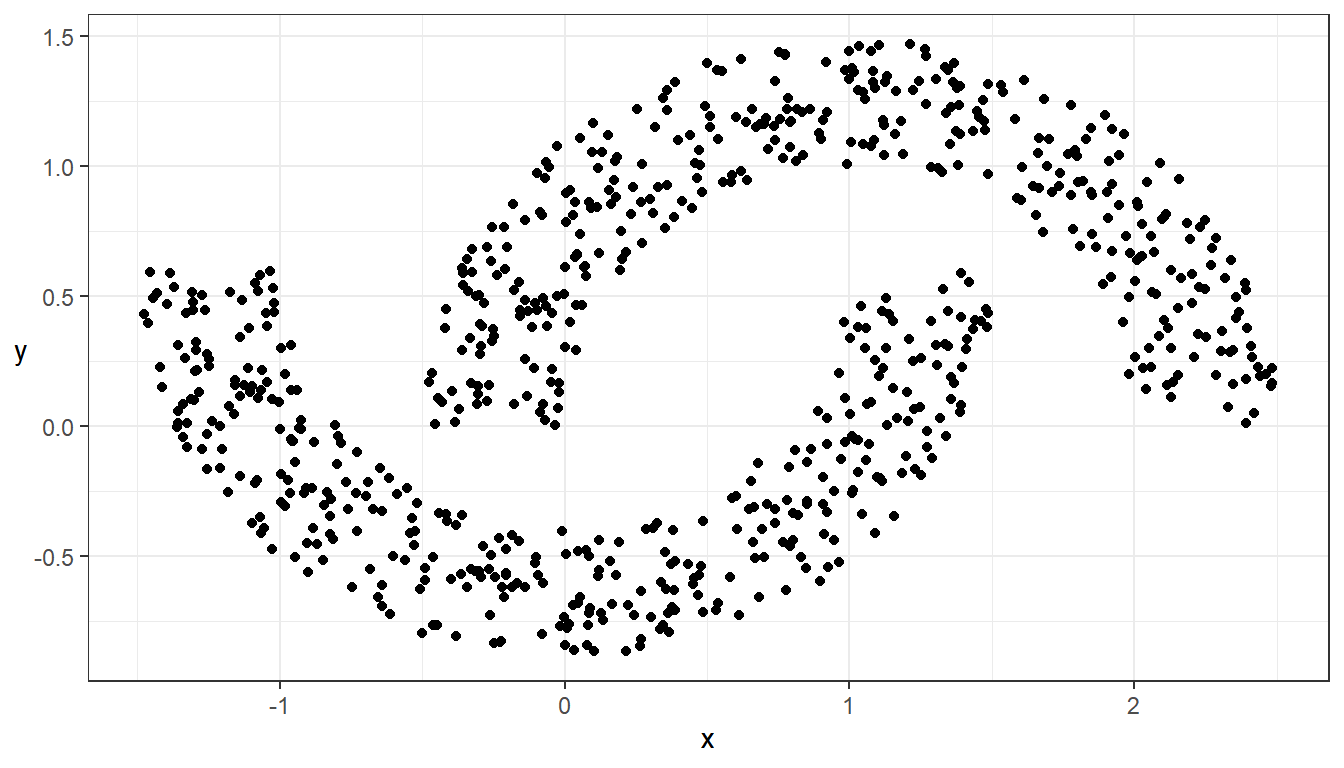
## Visualisera data
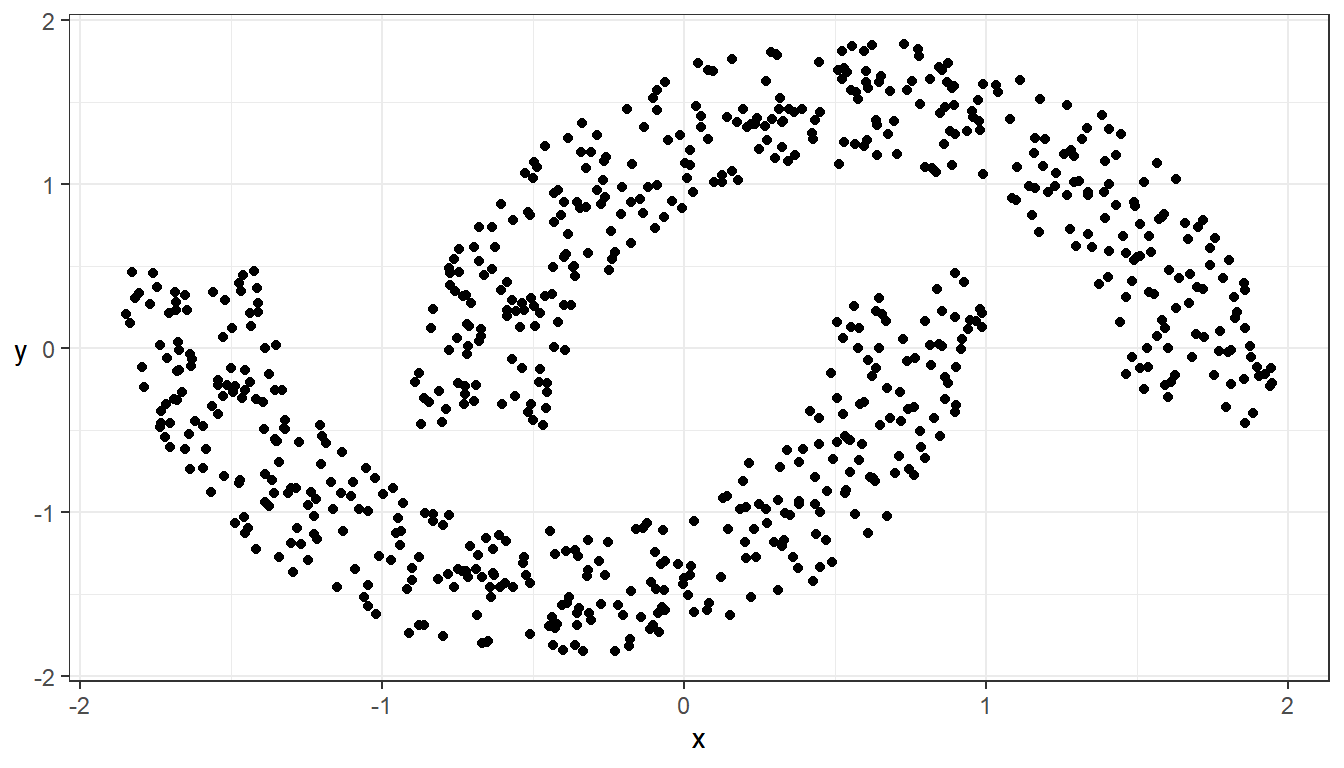
ggplot(data = data_clust_arbi, aes(x = x, y = y)) + geom_point() + theme_bw() +
theme(axis.title.y = element_text(angle = 0, vjust = 0.5))
## STANDARDISERA
data_clust_arbi_std <- data.frame(scale(data_clust_arbi, center = TRUE, scale = TRUE))
ggplot(data = data_clust_arbi_std, aes(x = x, y = y)) + geom_point() + theme_bw() +
theme(axis.title.y = element_text(angle = 0, vjust = 0.5))
## Skattar Agglomerativ Hierarkisk Klustring (AGNES)
cluster_results <-
agnes(
x = data_clust_arbi_std,
# Anger distansmåttet som ska användas. Kan också ange Manhattan
metric = "euclidian",
# Här anges vilken länkningsmetod som ska användas (ward, complete, average, single)
method = "single"
)
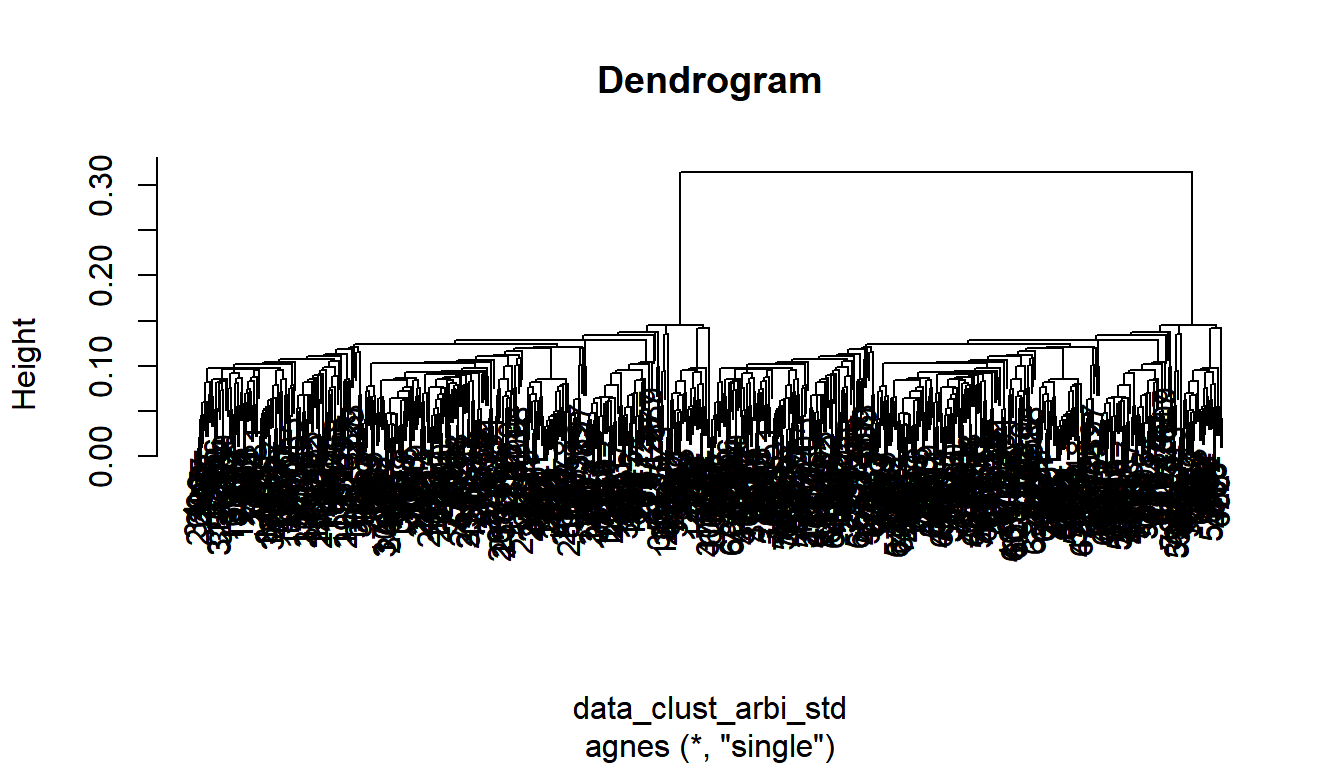
För att utvärdera resultatet av agnes() krävs en visualisering av alla ihopslagningar genom ett s.k. dendrogram. Nedan följer kod som visar visualiseringen av dendrogrammet och hur vi plockar ut ett manuellt angivet antal kluster från detta. Dendrogrammet ska påvisa ett “stort” avstånd mellan två intilliggande ihopslagningar för att vi ska anse att algoritmen hittat en bra uppdelning.
## Visualiserar resultatet i ett dendrogram
pltree(cluster_results, main = "Dendrogram")
## Anger hur många kluster som syns i dendrogrammet
clusters <-
cutree(
# Resultatet av den hierarkiska klustringen
cluster_results,
# Antalet kluster som enligt dendrogrammet bör vara då vi avslutar algoritmen
k = 2
)
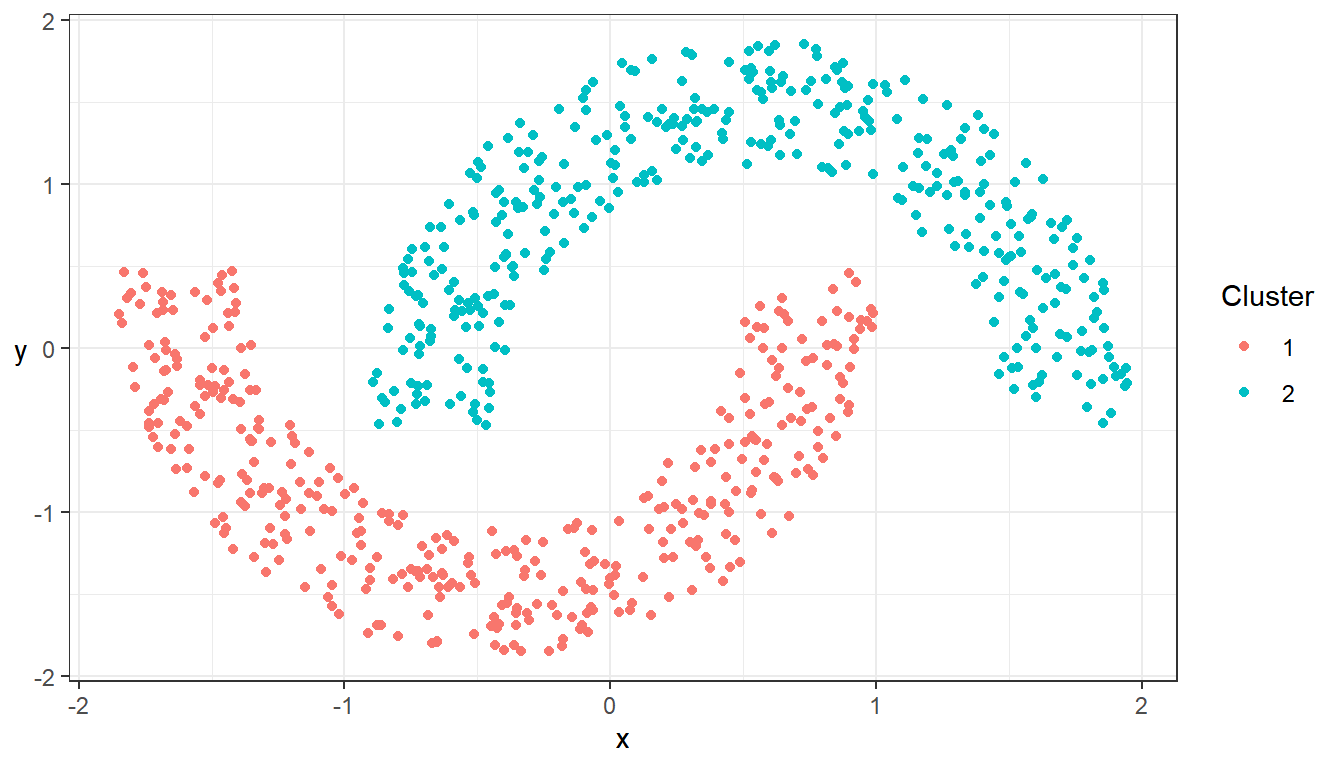
## Visualiserar resultatet
ggplot(data = data_clust_arbi_std,
aes(x = x, y = y)) +
geom_point(aes(color = factor(clusters))) +
theme_bw() +
theme(axis.title.y = element_text(angle = 0,
vjust = 0.5)) +
scale_color_discrete(name = "Cluster")
Täthetsbaserad klustring
Täthetsbaserad klustring försöker titta på en alternativ form av defintion av kluster. För dessa algoritmer baseras ett kluster på områden av högre täther separerade av områden med lägre täthet. En fördel med denna definition är att vi kan hitta mer godtyckliga former och kan ta hänsyn till brus i data på ett helt annat sätt.
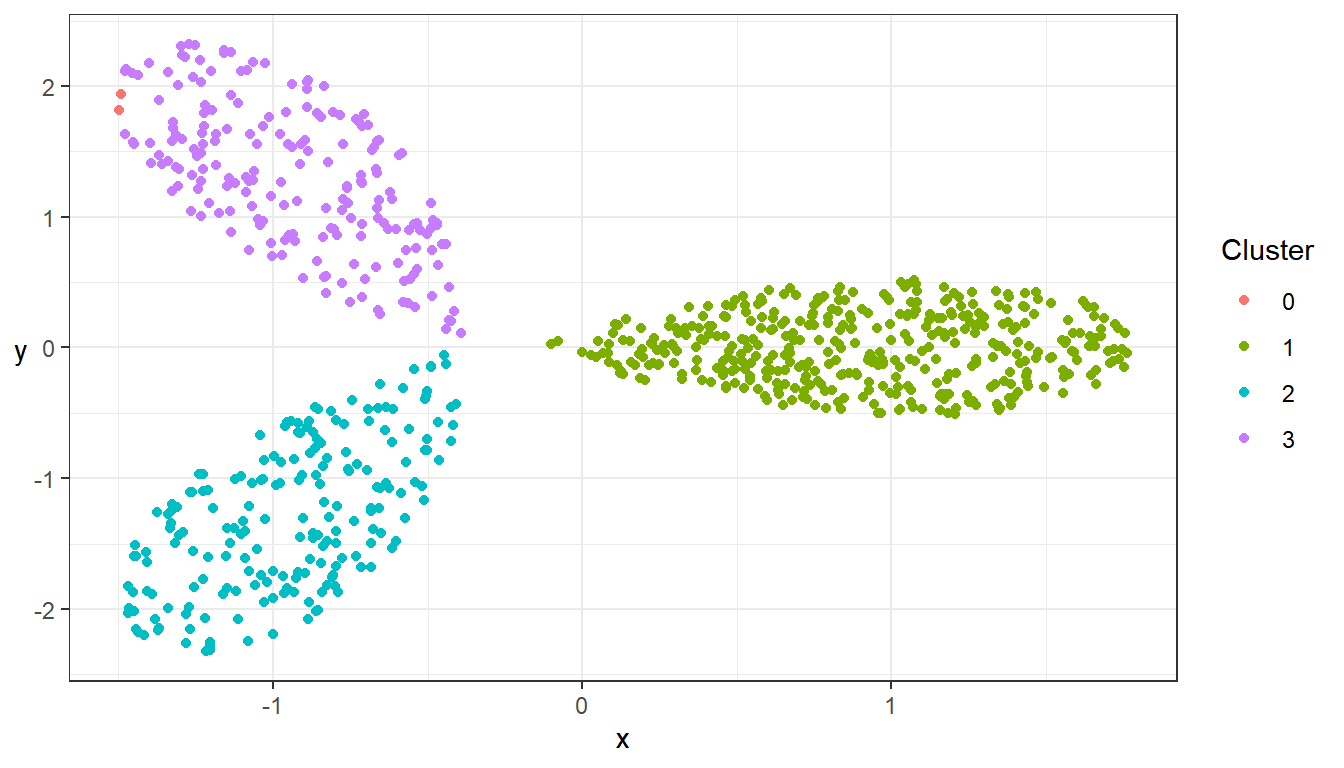
Exempelvis kan DBSCAN identifiera kluster, men också brus (som anges som klustertillhörighet 0).
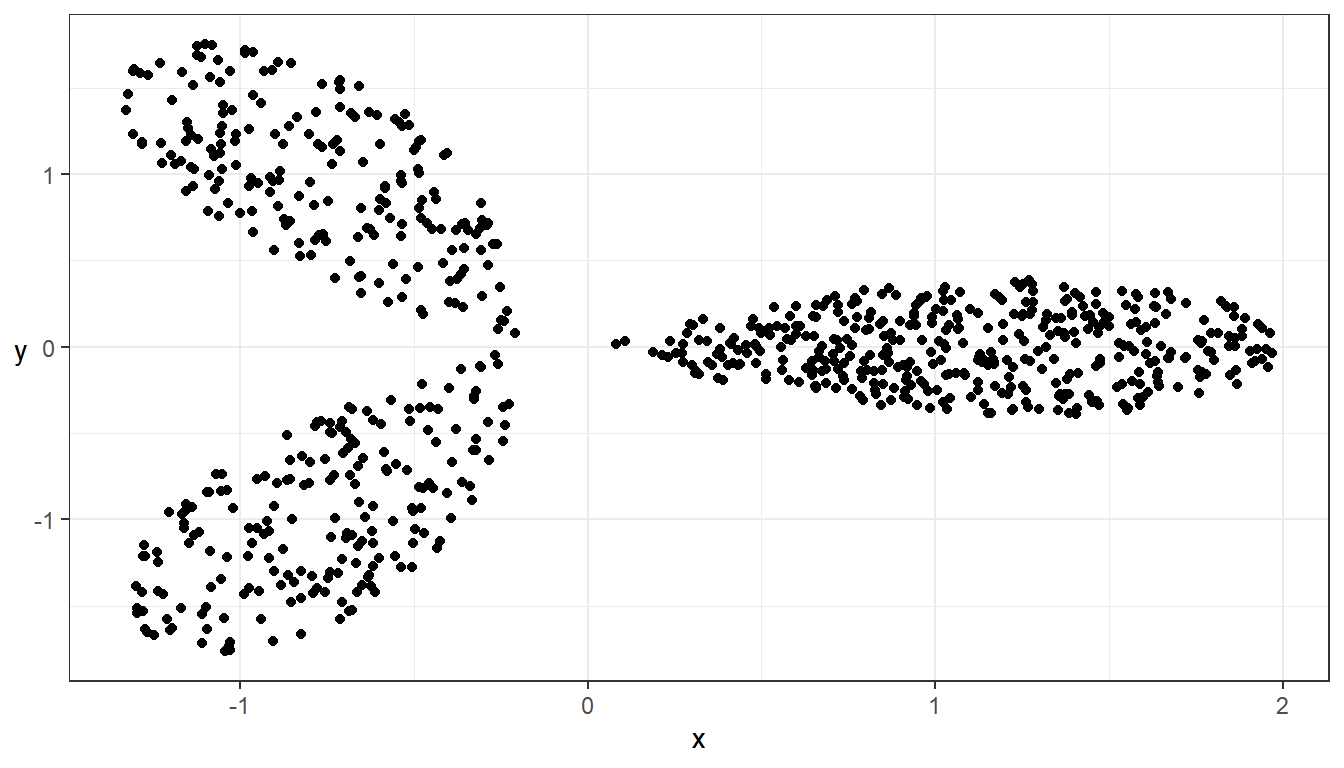
## Visualisera data
ggplot(data = data_clust_flow, aes(x = x, y = y)) + geom_point() + theme_bw() +
theme(axis.title.y = element_text(angle = 0, vjust = 0.5))
## STANDARDISERA
data_clust_flow_std <- data.frame(scale(data_clust_flow, center = TRUE, scale = TRUE))
ggplot(data = data_clust_flow_std, aes(x = x, y = y)) + geom_point() + theme_bw() +
theme(axis.title.y = element_text(angle = 0, vjust = 0.5))
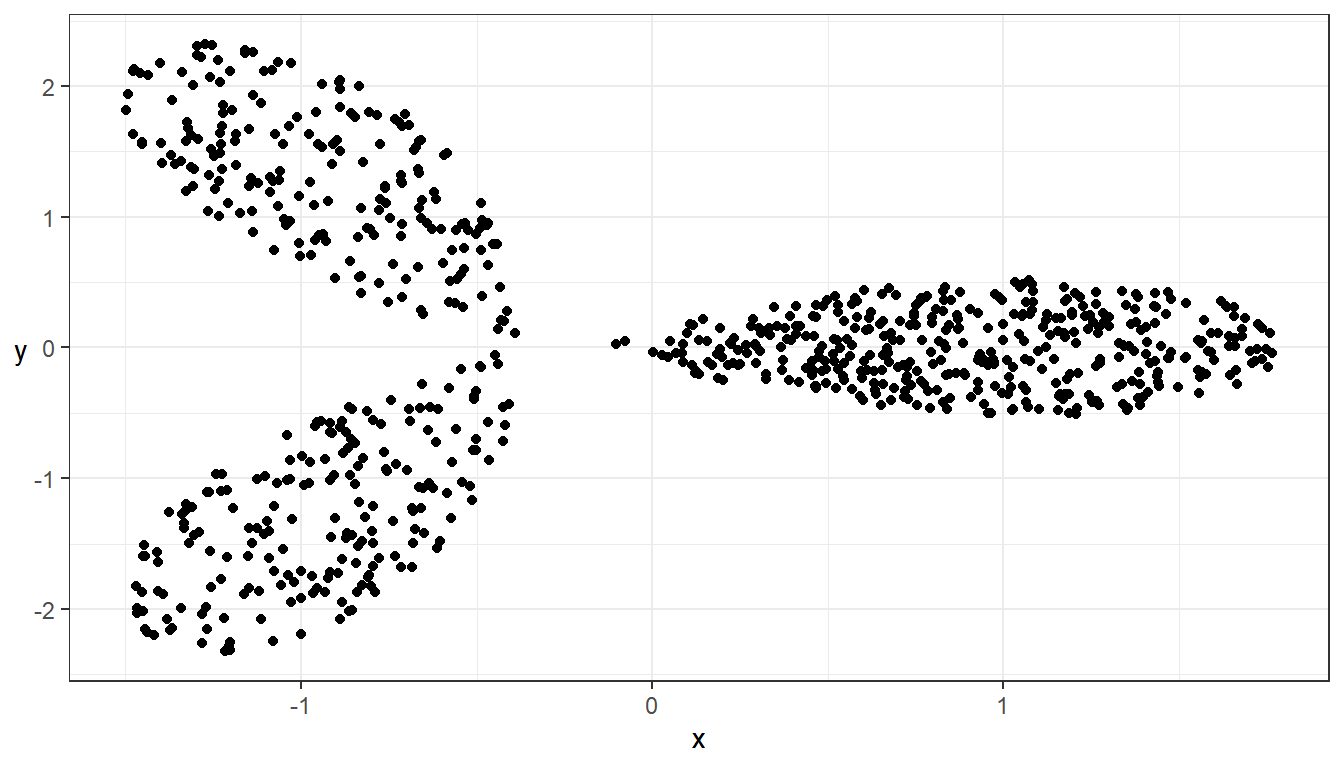
## Klustra med hjälp av DBSCAN
cluster_results <-
dbscan(
data_clust_flow_std,
# Anger sökradien runt en observation
eps = 0.15,
# Anger hur många punkter som måste finnas inom eps för att räknas som en kärnpunkt
minPts = 5,
# Anger att gränspunkter ska tilldelas ett kluster, annars räknas de som brus
borderPoints = TRUE
)
## Visualiserar resultatet
ggplot(data = data_clust_flow_std, aes(x = x, y = y)) +
geom_point(aes(color = factor(cluster_results$cluster))) + theme_bw() +
theme(axis.title.y = element_text(angle = 0, vjust = 0.5)) +
scale_color_discrete(name = "Cluster")
SOM/Kohonen kartor
En klustringsmetod som tar hänsyn till sambandet (närheten) mellan klustercentroider är SOM/Kohonen kartor.
Nedan följer lite setup-kod för SOM-exemplet.
## Laddar och standardiserar data
data(wines)
wines <- scale(wines)[,1:6]
## Funktion som byter håll på färgerna i inbyggda heat.colors funktionen
heat_colors_rev <- function(n, alpha = 1) {
rev(heat.colors(n, alpha))
}
Dessa kartor skapas i R med funktionen som() från paketet kohonen.
## Skattar kartan, beräknar centroiderna
set.seed(129)
results <-
som(
X = wines,
# Anger arkitekturen för kartans galler
grid =
somgrid(
# Anger dimensionerna för gallret
xdim = 3,
ydim = 4,
# Anger att vi vill ha en rektangulär topologi (4 grannar/centroid)
topo = "rectangular",
# Anger hur vi vill ta hänsyn till observationernas avstånd (kernel)
neighbourhood.fct = "bubble"
)
)
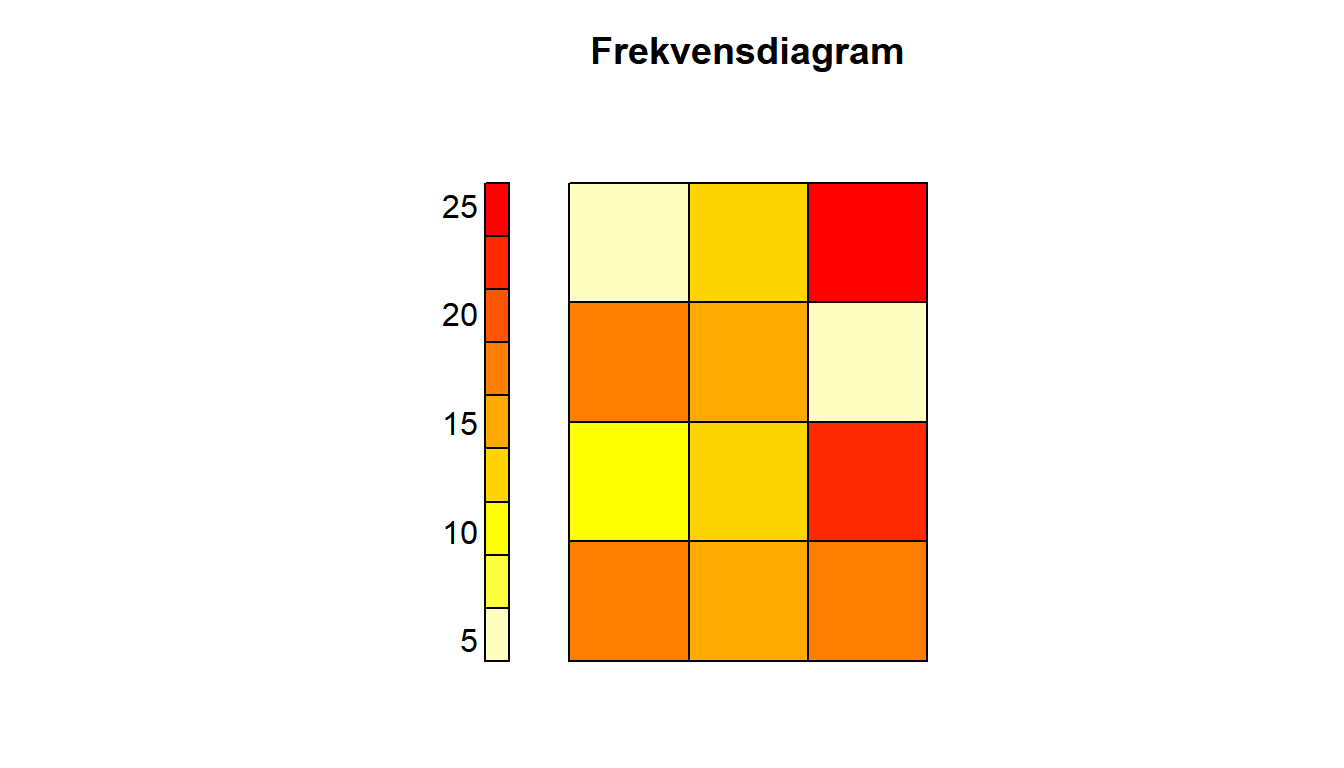
För visualisering av de resulterande gallret används plot.kohonen(). För visualiseringen av klusterresultatet kan splom() från lattice-paketet användas. Den skapar då en punktdiagramsmatris för alla variabler och de tilldelade klustren från $unit.classif.
## Visualiserar centroidernas positioner gentemot varandra, plot.kohonen för dokumentation
plot(
# Klusterresultatet
results,
# Anger vad färgerna i diagrammet ska visa
type = "counts",
# Anger raka kanter i kartan
shape = "straight",
# Anger en färgpalett
palette.name = heat_colors_rev,
# Anger en titel på diagrammet
main = "Frekvensdiagram"
)
## Om endast två dimensioner används kan liknande visualisering som tidigare klustringar användas
splom(
x = wines,
# Klasserna som ska färgläggas
groups = results$unit.classif,
# Tar bort axelvärden
pscales = 0,
# Anger att en liten fylld cirkel ska användas som symbol
pch = 20,
# Anger opaciteten (lägre opacitet möjliggör att se områden med överlappande observationer enklare)
alpha = 0.6
)
